
摘 要 肽作为重要的生理活性物质一直受到相关领域的广泛关注。 近年来,由于肽在细胞信号转导中所扮演的中心角色以及作为生物药物靶向蛋白质相互作用网络等特殊性质的发现,再次唤起了人们对肽的浓厚兴趣。 与之相伴的是,肽的理论和计算研究工作快速增长,并取得了长足进展。本文以"计算肽学"为主题系统概括了该领域的研究范畴和研究特点,并分别从肽的数据库构建、功能活性预测、分子对接、动力学模拟、结构数据分析、分子设计修饰以及系统生物学行为等几方面分类介绍了计算肽学的主要研究方向和当前发展状况。重点在于探讨采用计算化学和生物信息学方法剖析肽与蛋白质识别和相互作用的分子机制和理化基础,进而为肽类药物设计提供理论指导。此外,本文还提出了计算肽学在肽类纳米材料及生物表面活性剂等领域的潜在应用前景。
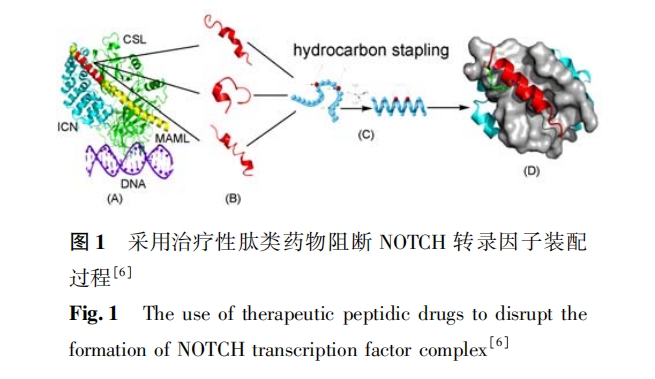
本综述分为 3 个部分 ,其内容安排如下:(1)引 言部分介绍肽学诞生的学科背景以及在此背景中形成的计算肽学这个分支领域 ,作为提引;(2)研究方向部分分类探讨计算肽学的研究内容和最新进展 , 作为主体;(3)总结及展望部分针对计算肽学的当前研究状况及未来发展趋势加以简要概括 , 作为收尾
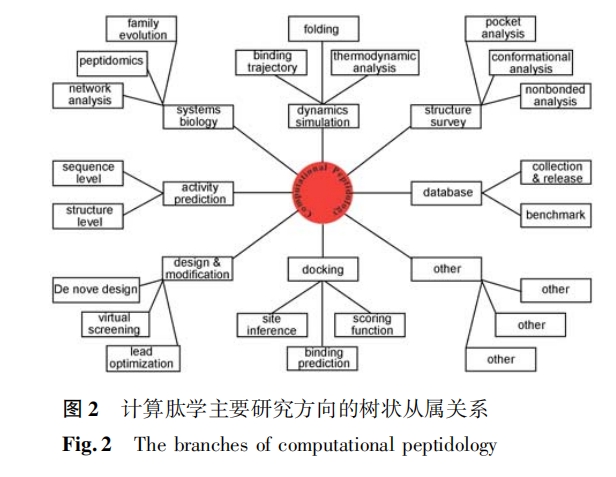
2 计算肽学的研究方向
迄今科学共同体总共发布了几十个肽相关数据库 ,其中多数是针对特定目的建立起来的肽序列-功能库 ,如免疫表位库 IEDB[23] 、抗菌肽库 APD[24] 、功能食品肽库 BIOPEP[25] 以及综合性的活性肽数据库 PepBank [26] 和 BioPD[27] 。这些资源对于研究肽的一 级序列模式与特定活性功能关系带来了极大便利 , 也为生物信息学家发展机器学习模型提供了丰富的数据来源 。如 IEDB 数据库已成为当前免疫信息学研究的公共数据平台 ,人们利用其广泛开展了抗原肽分析和肽疫苗设计等方面的研究。
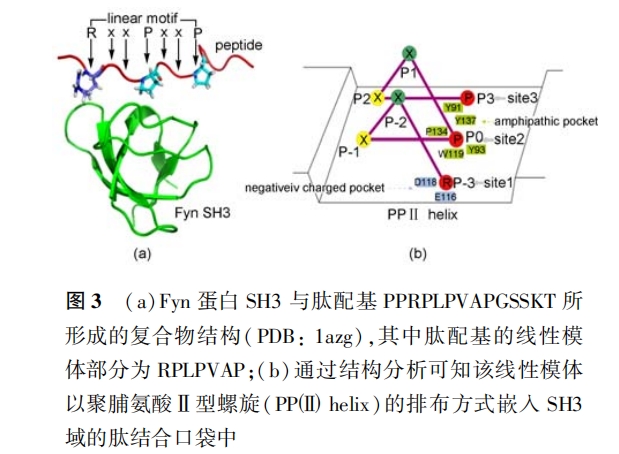
更高一个层次的原子结构水平 , 目前主要存在两个综合性的蛋白质/肽复合物数据库 , 即 3did[35] 和 Pepx[36] ;它们从生物大分子结构数据库 PDB[37] 中提取蛋白质(或结构域)与肽形成复合物的空间结构数据并加以归类注释 ,如通过对比蛋白质表面肽结合口袋的空间构造而对复合物进行分类 。这类数据库已被广泛用于蛋白质/肽相互作用结构信息 分析和提取(参见 2. 5 节)。遗憾的是 ,这些结构数据库并未提供相应活性或亲合力信息 。一般认为 , 要想全面深入研究生物分子识别机制 , 除了获知复合物的三维结构以外还需了解该结构对应的生物活性值 。因此 ,可以预料 ,构建结构-活性关联数据库将成为肽数据库未来发展方向之一。
精确可靠的计算识别和预测生物活性不仅是对肽而且对其他生物分子而言都是至关重要的 , 因为这是高通量虚拟筛选和理性分子设计的基础 。目前 ,肽的功能识别及活性预测主要在基于序列和基于结构两个方面展开 。前者仅利用肽的一级序列信息 ,采用统计建模方法 ,与相应生物活性加以函数关 联 ,建立预测模型;后者则挖掘已知的蛋白质/肽复合物结构信息 , 并从空间原子层次预测肽的生物活性。
(1) 序列层次 : 在定性水平 ,采用日益丰富的 SLiM 知识推测蛋白质功能位点成为常用策略。该法由于其简单高效而被广泛用于在基因组水平预测 信号肽、酶切位点、磷酸化区域和肽配基 [38] 。更进一步 ,生物信息学家使用大量已知功能肽序列训练机器学习工具( 如人工神经网络和支持向量机),继而用于评估未知样本 [39] 。在定量水平 ,化学计量学家发展起来的定量构效关系(QSAR)成为预测肽生物活性的常见手段[40] 。Hellberg 等早期的研究奠定了该法基本框架 ,其思路为 [41] : 收集氨基酸的大量性质参数;采用主成分分析(PCA)提取这些参数中所隐含的显著信息得分 , 称为主性质(principal properties);使用这些少量主性质代替大量原始变量 , 获 得 所 谓 的 氨 基 酸 描 述 子( amino acid descriptors)可用以参数化肽的一级序列结构;进而 采用偏最小二乘( PLS)等回归技术统计关联肽的结构参数与生物活性 ,获得的函数关系可用于预测目的。 常见氨基酸描述子包括 Z 标度[41] 、ISA-ECI 指 数 [42] 、VHSE 得分[43] 等。该法的缺点是无法处理长度不一的肽序列。虽然后来有人提出了诸如自交叉协方差( auto-cross covariance, ACC)[44] 等方法加以解决 ,但因物理意义不甚明确而未得到广泛应用。
(2)结构层次 : 由于结构数据大大少于序列数据 , 因此基于结构预测肽的生物活性还鲜见报道 ,但它具有许多相对于基于序列预测方法所不可比拟的 优势 ,如结果物理化学意义明确、所获知识易于指导结构改造等。以往 ,基于结构预测肽活性研究最多的对象是主要组织相容性复合物(MHC)[45] , 它是重要的细胞免疫相关蛋白 ,专司负责胞内肽抗原加工提呈 ,对疫苗设计意义重大。因此现有大量晶体结构数据可供利用 [46] 。另外 ,域/肽相互作用也是 基于结构预测肽活性的关注热点 ,主要研究对象包 括 SH3 域、WW 域、PDZ 域等。如 Hou 等采用同源模建、分子对接及动力学模拟在结构水平揭示了人类双载蛋白 SH3 域( hAmph SH3 )与配基肽的作用模式 ,并在此基础上建立了用于肽亲合力预测的三维定量构效关系(3D-QSAR)模型[47] 。 之后他们又提出分子相互作用能分解与支持向量机联用方案 (MIEC-SVM )筛 选 基 因 组 中 的 潜 在 SH3 肽 配 基 [48,49] ,所得结果得到了肽阵列( peptide array)实 验确 认 [50,51] 。 最近 , 我 们将量子力学/分子力学 (QM/MM)杂化计算用于提高 OppA 及 PSD95 蛋白 质与寡肽亲合力预测的精度 ,亦取得了成功 [15,16]。然而 , 这些研究都是针对特定肽/蛋白质体系开展的 ,所获得的预测模型仅适用于特定问题 ,不具通用性。Woo 和 Roux 曾发表了一套严格的肽/蛋白质结合自由能计算理论 ,但因需长程动力学模拟和复杂热力学路径分解而限制了该法的推广 [52]。
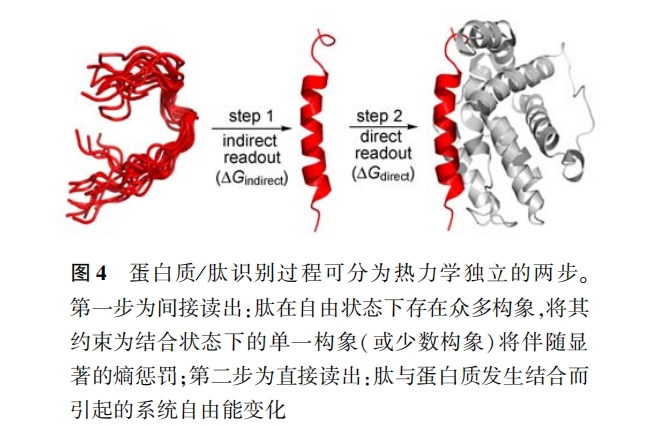
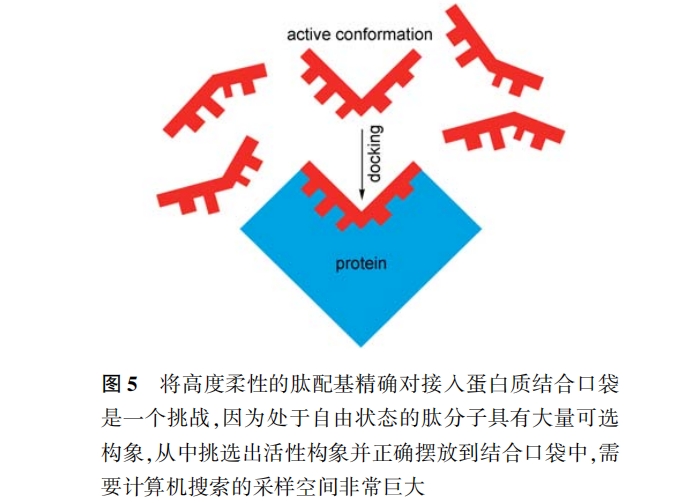
鉴于以上原因 , 已有部分研究者开始尝试发展专门的肽对接方法 。早期人们提出采用启发式策略和智能算法实现肽对接 [59] ,但缺乏全面测试和深入评价而未得到广泛应用 。后来多数研究者集中于采用分子动力学/Monte carlo/模拟退火采样与结构优化相结合的方法处理蛋白质/肽结合问题 ,提出了一系列肽对接方法 , 如 Gδ [60] 、Docscheme [61] 、 DynaDock [62] 等 , 特 别 是以色列希伯来大学的 schueler-Furman 及其合作者首次实现了肽对接的在线服务工具 FlexpepDock[63] ,测试表明该法对某些 体系可以达到“ 亚埃( sub-angstrom)”级的对接精度[64] 。最近,Donsky 等也发布了另一个在线肽对接工具 pepcrawler[65] ,为面向应用目的提供了极大便利 。此外 ,肽动态对接方案不仅用于预测肽在蛋白质活性口袋中的结合方式 ,还用于研究结合过程的动力学机制。Ahmad 等采用动力学对接首次在分子水平上全程模拟了 sH3 域与一个十肽的结合过 程 ,据此提出的双态模型包括一个初期快速弥散阶段和一个后期界面水分子排干过程;他们指出长程静电效应和短程疏水力分别是推动结合过程前后两个阶段发展的关键驱动因素 [66] 。该模型的整体轮廓与后来 staneva 和 wallin 利用全原子 Monte carlo 模拟 pDZ 域识别寡肽配基结论基本一致 ,后者也证实了域/肽结合过程存在两个分别由静电和疏水支配的阶段 [67] 。所不同的是 Ahmad 等认为第一阶段非常短暂且容易逾越 ,而 staneva 等却认为第一阶段是限速步骤 。有时当大量同类蛋白质/肽复合物结构已知的情况下(如上文提到的 MHc),对接问题可以简化为基于肽配基的公共主链构象预测目标侧链 ,然后再组合其他分子模拟方法对整体结构模型进行修正 , 这样可以 大大提高计算的效率和精度 [68]。
除此之外 , 目前针对肽对接的配套研究还非常缺乏 ,如发展专门的肽对接评分函数以及预测肽的结合位点等 。值得一提的是,Petsalaki 等基于平均 场论提出肽在蛋白质表面的结合位点预测方法是该 方面的重要进展 [69] 。
此外 ,肽的动力学模拟也常被用于肽对接意图 , 或用于精修肽对接所获得的初始结构;虽然此法效率较低 ,但是往往能够得到较为可靠的蛋白质/肽复 合物模型 , 因此亦常采用 [62] 。
随着 PDB 数据库[37] 中晶体学或多维 NMR 技术解析的肽与蛋白质所成复合物结构快速增长 ,从这些实验结构数据中提取和归纳有用知识为解释蛋 白质/肽识别和相互作用行为及预测潜在的作用模式提供了非常有价值的参考信息 [78] 。vanhee 等调 查了 731 个已知结构的蛋白质/肽界面后认为肽与 蛋白质 结合方式非常类似于单体蛋白的折叠模式[79] ,该发现为基于丰富蛋白结构数据设计蛋白 质/肽相 互作用提供了理论依据 。然而 , 稍 后 London 等进一步深入分析一组高质量样本后指出 , 肽往往采用比蛋白折叠更牢固的方式与受体结合 , 从而弥补伴随该过程可观的熵惩罚 [54] ;此外他们还发现一些有趣的现象 , 比如肽配基通常结合到蛋白质表面最大凹陷处 ,蛋白质/肽界面常存在关键性的 “ 热点残基( hotspot residue)”等 。另外一些研究者通过考察蛋白质复合物结构来研究肽调节的蛋白质相互作用 ,如 Jochim 和 Arora 通过对近万个多元蛋白复合物检视发现其中约有 13% 的界面包含螺旋模体 ,暗示了螺旋肽是非常有希望的蛋白复合体装 配的抑制构架 [80] 。的确 ,迄今实验确认的靶标活性肽多数为螺旋性 ,推测这可能是因该类二级结构稳定化程度较高且易于化学约束之故 [81] 。此后一些研究发现 ,除了螺旋模体之外 ,其他类型的线性序列 ( 如环状序列)也常出现于蛋白质复合物界面并对后者形成起到了重要的推动效应;由此估计肽段调节的蛋白质相互作用可多达 50% ,且它们都是潜在的蛋白质复合物装配“ 自抑制肽( self-inhibitory peptide)”[82] 。然而 ,上述结论需要考虑更多的因素才有意义 ,如 stein 等通过对大量肽调节的蛋白质相互作用进行拓扑能量分析后指出核心线性序列往往需要在特定的界面环境中才能保证高的特异性和适当的稳定性 ,环境因素平均贡献了 20% 以上的亲合 力[83] 。因此设计自抑制肽时如何包含界面环境贡 献是设计者需要考虑的主要因素之一。
理性设计特定肽分子并对其结构进行修饰改造 ,使之获得高的靶标亲合力和选择性以及良好的代谢稳定性和膜穿透能力是肽类药物研发者追求的梦想 。方法学方面 , 人们提出了Rosetta [84] 和 vitAL [85] 等方案进行全新( de novo)肽配基设计 ,而 我们也曾开发了 LigEvolutioner 程序用于已知先导肽( lead peptide)的结构自动进化改良[86] 。近年来 , 理性肽设计亦与各类生物活性检测实验紧密结合起来 ,这些工作成功地将理论推向了应用 。它们主要 在三个层次上得到实现 :一是基于序列知识 ,通过统计学习模型和生物信息学方法提炼和优化目标肽序列 ,如 Edwards 等筛选血小 板活性调节因子[87] , shemesh 等发现G蛋白偶联受体激动剂 [88]以及 walshe 等确定人类白细胞抗原结合表位[89] 等是该水平研究的典型代表;二是基于模糊结构信息 ,通过序列推测靶标蛋白的常见结构模架 ,则可根据已知的大量该类模架结构定义目标肽 ,如 kliger 等采用 Fourier 变换设计伴侣蛋白的活性调节肽[90] 及 yin等开发的整合素跨膜区靶向肽 [91] 。后者可视为该领域的突破性工作 , 因为研究者在完全未知整合素跨膜结构的情况下仅利用已知少数膜蛋白的跨膜螺旋空间排列模式就设计出了能够自动装配到细胞膜内并与整合素靶标区域发生特异性作用的活性肽;三是基于精确靶标结构数据 ,通过分子模拟方案和能量分析策略获得高性能的肽配基 ,如 cui 等发现促肿瘤凋亡肽 [92] ,sood 构造肿瘤蛋白调节肽[93] 以及 Grigoryan 等设计亮氨酸拉链(bZIP)特异攻击肽 [94] 。后一个工作特别有趣 , 因为作者首次报道了定向设计肽的特异性(而非亲合力)的成功案例。
相对上述特定案例分析而言 ,系统生物学研究肽的性质和活性更着眼于在整个基因组水平、整个细胞网络和整个家族分类中考察肽与靶标之间的复杂作用行为和多维相互关系 [95] 。一般采用高通量实验手段 , 如噬菌体展示肽库[96] 和 sPOT 合成技术 [97] ,可以在短时间内产生大量的候选肽 ,进而筛选它们对特定靶标( 或家族靶标)的亲合潜力[98] 。这些数据是重要的系统肽生物学研究资源 。而基于序列和结构产生的理论模型亦被用于构造特定靶标的完整基因组识别剖面 [99,100] 。此外 ,从序列、结构和作用对象角度探讨某一类肽识别域的家族内部关联性和个体间差异性亦是在系统水平研究域/肽分子进化的常见策略 [101,102] 。对此 , 这里仅提及 stiffler 等的工作[103] :他们采用亲合力检测和主成分聚类相结合的办法研究了小鼠体内表达的 157 种PDZ 域在配基选择性空间的分布情况 ,结果表明 ,整个PDZ 域家族通过进化优化后呈均匀分散在该空间中 ,从而最大限度避免了个体之间的交叉反应 ( cross-reactivity )。 近年来兴起的肽 组学(peptidomics)以及在此基础上发展起来的肽组学库和数据挖掘技术可谓后起之秀 ,必将在未来大放异彩 [104,105] 。
3 总结及展望
随着高通量测序技术和系统组学方法的高速发展,kahvejian 等在其前瞻性论著中预测 ,不出 20 年人们将测定几乎所有的生物学数据 — 至少可以随意测定自己想要的数据 [106] 。从而 , 多数生物学家都(或多或少)逐渐转变成为计算生物学家 , 因为那时生物学家的主要精力会用于利用计算工具从海量数据中发掘生物学知识 — 正如 Jorgensen 宣称的那样 [107] :we, re all computational biologists! 尽管这样的论断过于激进 ,但向人们昭示了理论和计算方法以及信息挖掘技术在生命及其相关学科中的广阔应用前景 。近年来 , 由于肽在细胞信号转导中所扮演的重要角色以及作为生物药物靶向蛋白质相互作用网络等特殊性质的发现 ,唤起了科学共同体对肽的浓厚兴趣 。与之相伴的是 ,计算化学和生物信息学领域与肽相关的研究工作日益增多 ,并取得了长足进展 。鉴于肽的理论计算研究开始起步 ,并已渐进佳境 ,本文以计算肽学为主题概括该领域的研究范畴和研究动态 ,其目的是希望将之系统化为一个专门分支学科便于相关研究者交流探讨 。可以预 见 ,计算肽学的发展必将受到计算科学和肽科学两方面进展的推动 ,今后其研究重点应该主要集中在下列几个方面:(1)随着蛋白质/肽复合物三维结构数据的日益累积 ,在原子水平考察肽与蛋白质识别和相互作用的理化基础及热力学性质将得到进一步发展。(2)采用分子设计方法获得高性能肽模拟物攻击蛋白质相互作用网络已开始崭露头角 ,未来有望成为新药研发的一个高速增长点。(3)构建肽组学数据库及挖掘内涵信息可在系统生物学层次阐释细胞信号转导及代谢通路中各类肽的功能与活性。(4)以肽为基础开发自组装纳米管、表面活性材料、 药物传输系统等相关实验研究已有报道 ,相信计算 模拟和理论设计工作将在不久之后广泛展开。